ChIP-exo - ChIP-exo
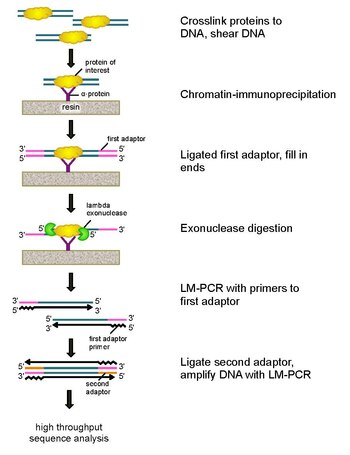
ChIP-exo a xromatin immunoprecipitatsiyasi qiziqish oqsillari joylashgan joylarni xaritalashga asoslangan usul (transkripsiya omili ) genom bilan bog'lanadi. Bu. Ning modifikatsiyasi ChIP-seq protokol, yuzlab saytlardan majburiy saytlarning o'lchamlarini yaxshilash tayanch juftliklari deyarli bitta asosiy juftlikka. Bu foydalanishni ishlatadi ekzonukleazalar 5'-3 'yo'nalishidagi oqsil bilan bog'langan DNKning zanjirlarini oqsillarni bog'lanish joyining oz sonli nukleotidlari ichiga tushirish. Ekzonukleaza bilan ishlangan uchlarining nukleotidlari ba'zi birikmalar yordamida aniqlanadi DNKning ketma-ketligi, mikroarraylar va PCR. Keyinchalik, ushbu ketma-ketliklar genomga joylashtirilgan bo'lib, oqsil bog'langan joyni aniqlash uchun.
Nazariya
Xromatin immunoprecipitatsiyasi (ChIP ) texnikasi 1984 yildan beri qo'llanilib kelinmoqda[1] protein-DNKning o'zaro ta'sirini aniqlash uchun. Natijalar sifatini yaxshilash uchun ChIP-da juda ko'p farqlar mavjud. Shunday yaxshilanishlardan biri, Chipdagi chip (ChIP-chip), ChIP-ni mikroarray texnologiyasi bilan birlashtiradi. Ushbu texnikada ayniqsa sezgirlik va o'ziga xoslik cheklangan jonli ravishda bu erda mikroraylovlar yadro bo'linmasida mavjud bo'lgan minglab oqsillar tomonidan cheklanib, natijada yuqori darajadagi noto'g'ri pozitivlar paydo bo'ladi.[2] Keyingi keldi ChIP ketma-ketligi (ChIP-seq), bu ChIPni yuqori o'tkazuvchanlik bilan tartiblashni birlashtiradi.[3] Shu bilan birga, kesilgan DNK fragmentlarining heterojen tabiati bog'lash joylarini ± 300 taglik juftlik oralig'ida xaritalab, o'ziga xosligini cheklaydi. Ikkinchidan, ifloslangan DNK jiddiy muammo tug'diradi, chunki juda oz miqdordagi genetik joylar qiziqish oqsili bilan o'zaro bog'liq bo'lib, har qanday o'ziga xos bo'lmagan genomik DNK fon shovqinining muhim manbasiga aylanadi.[4]
Ushbu muammolarni hal qilish uchun Ri va Pyu klassikani qayta ko'rib chiqdilar nukleazadan himoya qilish tahlili ChIP-exo dasturini ishlab chiqish.[5] Ushbu yangi ChIP texnikasi lambdaga tayanadi ekzonukleaz bu faqat 5b-3 ′ yo'nalishidagi bog'lanmagan ikki zanjirli DNKni va barchasini pasaytiradi. Qisqacha aytganda, qiziqish oqsili (epitop yorlig'i bilan muhandislik immunoprecipitatsiya uchun foydali bo'lishi mumkin) in vivo jonli ravishda formaldegid yordamida genom bo'ylab tabiiy bog'lanish joylari bilan o'zaro bog'lanadi. Keyin hujayralar to'planadi, sinib ochiladi va kromatin qirqib olinadi va eritiladi sonikatsiya. So'ngra o'zaro bog'liq bo'lgan DNK bilan bir qatorda qiziqish oqsilini immunoprecipitatsiyalash uchun antikor ishlatiladi. Keyin DNK PCR adapterlari uchlari bilan bog'lanadi, bu esa ekzonukleaza hazm bo'lgandan keyin ikkinchi zanjir DNK sintezi uchun boshlang'ich nuqtasi bo'lib xizmat qiladi. Lambda ekzonukleazasi keyinchalik 5 ′ uchidan DNKning ikki juft zanjirini hazm qilish oqsil-DNK kovalent o'zaro ta'sir chegarasida bloklanishigacha hazm qiladi. Aksariyat ifloslantiruvchi DNK ikkinchi darajali o'ziga xos ekzonukleaza qo'shilishi bilan parchalanadi. Keyin o'zaro bog'liqlik teskari yo'naltirilgan, PCR adapterlarining primerlari kengaytirilgan bo'lib, ikki qatorli DNK hosil bo'ladi va ikkinchi adapter 5 5 uchiga bog'lanib, ekzonukleazani hazm qilishni to'xtatish joyini aniq belgilaydi. Keyin kutubxona PCR yordamida kuchaytiriladi va mahsulotlar tomonidan aniqlanadi yuqori o'tkazuvchanlikni ketma-ketligi. Ushbu usul har qanday genom tarkibidagi har qanday oqsilni biriktirish joyi uchun bitta asosiy juftlikni echishga imkon beradi, bu ChIP-chip yoki ChIP-seqga qaraganda ancha yuqori aniqlik.
Afzalliklari
ChIP-exo oqsillarni biriktirish joylarini aniqlashda bitta asosiy juftlik rezolyutsiyasidan voz kechishi ko'rsatilgan. Bu ChIP-seqdan farqli o'laroq, u oqsilning bog'lanish joyini faqat ± 300 taglik juftlikda topishi mumkin.[4]
Oqsil bilan bog'lanmagan DNK bo'laklarining ifloslanishi ChIP tajribalarida yolg'on ijobiy va salbiy ko'rsatkichlarning yuqori bo'lishiga olib kelishi mumkin. Jarayonga ekzonukleazalar qo'shilishi nafaqat bog'lanish joyini chaqirishni yaxshilaydi, balki sekvensiya qilishdan oldin eritmadan ifloslangan DNKni olib tashlaydi.[4]
Nukleotid bo'lagi bilan samarasiz bog'langan oqsillarni ChIP-ekzo aniqlaydi. Bu, masalan, CTCF transkripsiyasi faktorini bog'lash joylarini ilgari topilganidan ko'ra ko'proq tanib olishga imkon berdi.[5]
Yuqori piksellar sonini va pasaytirilgan fon tufayli ChIP-exo-dan foydalanishda ketma-ketlikni qamrab olish chuqurligi kamroq bo'lishi kerak.[4]
Cheklovlar
Agar oqsil-DNK kompleksi bitta majburiy hodisa doirasida o'zaro bog'lanishning bir nechta joyiga ega bo'lsa, u holda bir nechta aniq bog'lanish hodisalari mavjud bo'lib ko'rinishi mumkin. Bu, ehtimol, ushbu oqsillarni xuddi shu hodisa doirasida mavjud bo'lgan majburiy joylardan birida denaturatsiyadan va o'zaro bog'lanishdan kelib chiqadi. Keyin ekzonukleaza bog'langan joylardan birida to'xtaydi, oqsil qaysi sayt bilan o'zaro bog'langaniga qarab.[5]
Ushbu texnikadan foydalanish uchun har qanday ChIP-ga asoslangan usulda bo'lgani kabi, qiziqish oqsili uchun mos antikor mavjud bo'lishi kerak.
Ilovalar
Ri va Pugh transkripsiya omillarining kichik to'plami bo'yicha tahlillarni o'tkazib ChIP-ekzo bilan tanishadilar: Reb1, Gal4, Phd1, xamirturushdagi Rap1 va odamda CTCF. Reb1 saytlari ko'pincha klasterlarda topilgan va bu klasterlar kutilganidan ~ 10 baravar yuqori bo'lgan. Klasterlardagi ikkilamchi joylar birlamchi bog'lanish joyidan ~ 40 bp. Gal4ning bog'lovchi motiflari to'rt nukleotidning uchtasiga kuchli ustunlik ko'rsatdi, bu Gal4 va chiqarib tashlangan nukleotid o'rtasidagi salbiy o'zaro ta'sirni ko'rsatdi. Phd1 uch xil motifni taniydi, bu esa Phd1 ning majburiy motifining noaniqligi to'g'risida oldingi xabarlarni tushuntiradi. Rap1 to'rtta motifni taniganligi aniqlandi. Ushbu oqsil bilan bog'langan ribozomal oqsil genlari ma'lum bir motifni kuchliroq konsensus ketma-ketligi bilan ishlatishga moyil edi. Boshqa genlar tez-tez kuchsizroq konsensusli motiflarning klasterlaridan foydalangan, ehtimol shunga o'xshash yashashga erishish uchun. CTCF ning majburiy motiflarida to'rtta "modul" ishlatilgan. Bog'langan CTCF saytlarining yarmi 1 va 2 modullaridan foydalangan, qolganlari esa to'rttasining kombinatsiyasidan foydalangan. Ushbu modullarning turli xil birikmalarini aniqlash uchun CTCF sink barmoqlaridan foydalanadi deb ishoniladi.[5]
Ri va Pyu boshlang'ichgacha bo'lgan kompleks (PIC) tuzilishini va tashkilotini tahlil qildilar Saxaromitsalar genomlar. ChIP-exo-dan foydalanib, ular boshqa kashfiyotlar qatorida TATA-bo'lmagan deb e'lon qilingan promouterlarning TATA o'xshash xususiyatlarini aniq aniqlashga muvaffaq bo'lishdi.[6]
Shuningdek qarang
Adabiyotlar
- ^ Gilmour, DS; JT Lis (1983). "Protein-DNKning o'zaro ta'sirini aniqlash jonli ravishda: RNK-polimerazaning spetsifik bakterial genlarga tarqalishi ". Milliy fanlar akademiyasi materiallari. 81 (14): 4275–4279. doi:10.1073 / pnas.81.14.4275. PMC 345570. PMID 6379641.
- ^ Albert, men; TN Mavrich; LP Tomsho; J Qi; SJ Zanton; SC Shuster; BF Pugh (2007). "H2A.Z nukleosomalarining translyatsion va rotatsion sozlamalari kesib o'tadi Saccharomyces cerevisiae genom ". Tabiat. 446 (7135): 572–576. Bibcode:2007 yil natur.446..572A. doi:10.1038 / nature05632. PMID 17392789. S2CID 4416890.
- ^ Ren, B; F Robert; Jey Urik; O Aparicio; EG Jennings; Men Simon; J Zaytlinger; J Shrayber; N Xannett; E Kan; va boshq. (2000). "DNKni bog'laydigan oqsillarning genom bo'ylab joylashishi va funktsiyasi". Ilm-fan. 290 (5500): 2306–2309. Bibcode:2000Sci ... 290.2306R. CiteSeerX 10.1.1.123.6772. doi:10.1126 / science.290.5500.2306. PMID 11125145.
- ^ a b v d Pugh, Benjamin. "Protein-nuklein kislotalarning o'zaro ta'sirini aniqlash usullari, tizimlari va to'plamlari". Amerika Qo'shma Shtatlarining arizalarini nashr etish. Amerika Qo'shma Shtatlari Patentlari. Olingan 17 fevral 2012.
- ^ a b v d Ri, Xo Sung; BJ Pugh (2011). "Bir nukleotidli rezolyutsiyada aniqlangan genom bo'yicha oqsil-DNKning o'zaro ta'sirlari". Hujayra. 147 (6): 1408–1419. doi:10.1016 / j.cell.2011.11.013. PMC 3243364. PMID 22153082.
- ^ Ri, Xo Sung; BJ Pugh (2012). "Eukaryotik boshlang'ichgacha bo'lgan komplekslarning genomik tuzilishi va tashkil etilishi". Tabiat. 483 (7389): 295–301. Bibcode:2012 yil natur.483..295R. doi:10.1038 / tabiat 1077. PMC 3306527. PMID 22258509.
Tashqi havolalar
- DNK-oqsilning o'zaro ta'siri yuqori aniqlikda
- Transkripsiya faktorining majburiyligini hal qilish
- Yuqori aniqlikdagi xromatin immunoprecipitatsiyasi
- Yangi usul bilan aniqlangan muhim gen-regulyatsiya oqsillari
- CexoR: ChIP-ekzo takrorlanishida yuqori aniqlikdagi protein-DNK o'zaro ta'sirini ochish uchun R / Biyo o'tkazgich to'plami
- Peconic Genomics
